Identyfikacja białek.
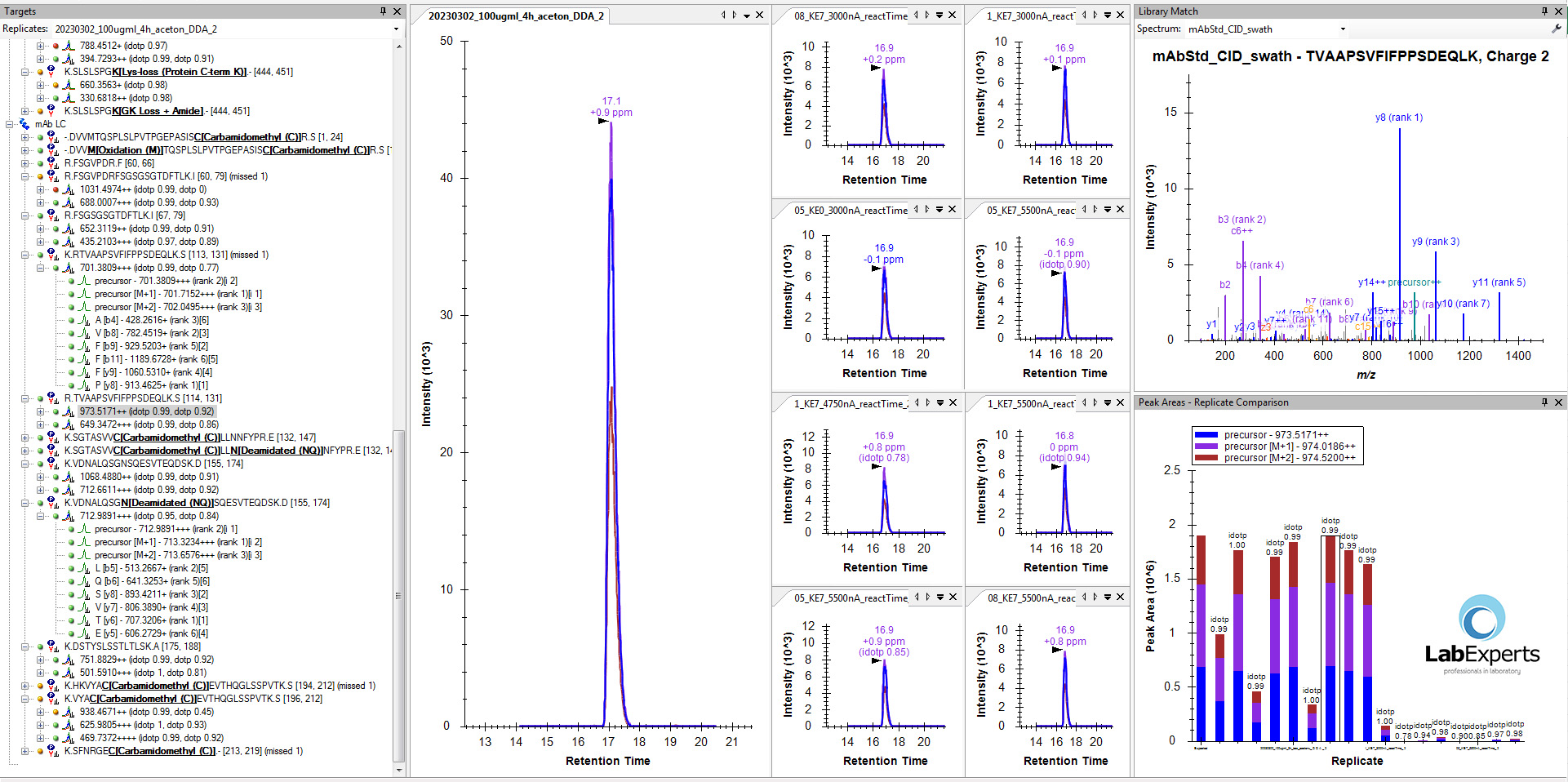
Identyfikacja białek opiera się na eksperymentalnym pomiarze mas cząsteczkowych peptydów oraz dopasowaniu sekwencji aminokwasów badanego białka do sekwencji białkowych zdeponowanych w bibliotekach (bazach danych). W badaniach proteomicznych najczęściej stosuje się dwa podejścia: top-down i bottom-up. Metoda top-down polega na analizie pełnych sekwencji białek. Z kolei analizy bottom-up wykorzystują trawienie białek enzymami proteolitycznymi. Otrzymane w wyniku trawienia peptydy analizuje się za pomocą spektrometru masowego. Każda analiza prowadzi do porównania danych eksperymentalnych z danymi uzyskanymi w wyniku teoretycznego trawienia białek opisanych w bazie danych.
LC-MS/MS
W celu określania sekwencji białkowych powszechnie stosuje się tandemowe spektrometry mas. Pozwalają one na selekcję jonów pseudomolekularnych peptydów a następnie ich fragmentację i pomiar mas powstałych fragmentów. Fragmentacja z użyciem dysocjacji indukowanej kolizją (CID – ang. Collision induced dissociation) jest nieocenionym narzędziem w celowanej i niecelowanej analityce MS/MS małych cząsteczek. Należy pamiętać, że w analizie proteomicznej dane uzyskane za pomocą wspomnianej metody fragmentacji mogą pozostawiać luki. Przykładowo, widma uzyskane w wyniku fragmentacji CID nie pozwalają na jednoznaczne różnicowanie niektórych aminokwasów: leucyny od izoleucyny (identyczne masy molowe i fragmentacja CID) oraz lizyny od glutaminy (bardzo podobne masy). Z tego powodu analizy proteomiczne wykonujemy na nowoczesnym wysokorozdzielczym urządzeniu ZenoTOF 7600 (SCIEX), w którym pułapka jonowa Zeno i technologia EAD (ang. Electron-Activated Dissociation) stanowią razem potężną kombinację. Umożliwia ona tworzenie unikalnych fragmentów, pozwalających na różnicowanie problematycznych aminokwasów oraz zwiększenie czułości i specyficzności analizy.
Wykonujemy analizy:
- prób z roztworu;
- prób z żelu;
- prób ze złożonej matrycy (po wcześniejszym ustaleniu);
- prób z liofilizatu;
- prób uprzednio przygotowanych przez klienta.
Analiza jakościowa białek obejmuje:
- W celu potwierdzenia obecności białka próbę poddaje się trawieniu i wynik otrzymany na podstawie danych eksperymentalnych jest porównywany z danymi teoretycznymi;
- Porównanie eksperymentalnych danych dotyczących mas peptydów wchodzących w skład analizowanego białka z sekwencjami białek opisanymi w bazie danych;
- Analizę i identyfikację mieszaniny białek;
- Identyfikację modyfikacji potranslacyjnych (ang. PTM – post translational modifications) (korzystamy z narzędzi: Skyline, Spectronaut)
- Raportowanie wyniku zawierające m.in. potwierdzenie pokrycia sekwencji białkowej uzyskanej w próbie badanej z białkiem teoretycznym oraz listę zmodyfikowanych peptydów, lokalizację oraz % modyfikacji.
Analizy porównawcze obejmują:
- Określenie składu mieszanin;
- Porównanie i profilowanie serii prób – identyfikacja białek i analiza trendu.
W naszej ofercie można również znaleźć:
- Wsparcie w planowaniu oraz wykonaniu badań (zakres ustalany jest indywidualnie);
- Pomoc w opracowaniu metod ekstrakcji ze złożonych matryc – płyny biologiczne, tkanka roślinna, tkanki zwierzęce (zakres ustalany jest indywidualnie);
- Analizę statystyczna za pomocą narzędzi: MarkerView (PCA), GraphPad Prism, Trendy, Sciex OS;
- Analizę ilościową białek;
- Analizę i charakterystykę przeciwciał monoklonalnych.